蛋白质序列比对是分子生物学和生物信息学中不可或缺的一步,尤其在研究同源蛋白、功能保守区域、变异位点或构建系统发育树时显得尤为关键。DNAMAN作为一款经典的序列分析工具,凭借其稳定性、操作简单和可视化友好的特点,广受实验人员欢迎。在具体应用中,DNAMAN如何比对蛋白、DNAMAN怎么多重比对结果查看和分析,这两个问题时常被问及。本文将围绕这两个主题展开详细讲解,并辅以实际操作建议与可视化技巧,助力用户高效进行蛋白序列比对分析。
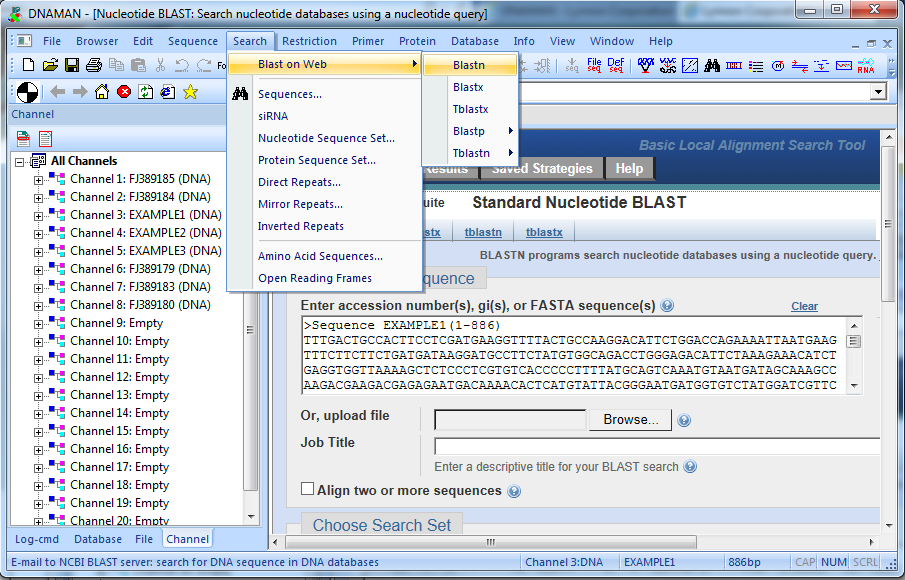
一、DNAMAN如何比对蛋白
DNAMAN支持多种序列类型的比对,包括DNA、RNA和蛋白质序列。其中蛋白质比对可以揭示序列之间的保守性、结构相似性和进化关系,尤其适用于研究不同物种之间的功能性蛋白同源序列。
1.导入蛋白质序列文件
打开DNAMAN软件;
点击菜单栏File→Import→Sequence;
选择本地的蛋白质序列文件(支持FASTA、TXT等格式);
多条蛋白序列可存储在一个FASTA文件中,每条以“>”开头标识。
导入后,所有蛋白序列将在左侧Workspace或窗口中逐条显示。
2.启动比对功能
多选你要进行比对的蛋白序列(按住Ctrl或Shift键);
点击菜单栏Align→MultipleSequenceAlignment(MSA);
在弹出选项中选择ProteinSequences;
软件会自动识别为氨基酸序列比对,并打开比对参数设置界面。
3.设置比对参数
算法选择:一般使用默认的ClustalW或Muscle;
打分矩阵:常用BLOSUM62、BLOSUM80、PAM250等;
Gappenalties(空位惩罚):可适当调整以优化比对质量;
设置完成后,点击“Align”开始比对。
4.查看比对结果
比对完成后,DNAMAN会展示对齐结果窗口;
每条蛋白序列占据一行,氨基酸对齐列显示;
保守位点自动高亮,可看到“*”、“:”或“.”等符号标注保守程度;
页面底部或右侧会有统计数据,如保守性得分、平均相似性等。
二、DNAMAN怎么多重比对结果
完成蛋白质比对之后,进一步的结果分析是关键,包括查看保守区、功能域注释、可视化导出等。DNAMAN提供多种方式对多重比对结果进行呈现和操作。
1.比对结果查看方式
DNAMAN提供几种对齐结果视图:
标准比对视图:横向展示所有序列对齐,每列为对应位点;
保守性视图:突出保守位点及其相对得分;
颜色编码视图:根据氨基酸理化性质(如疏水性、极性等)设置颜色,高度直观;
一致性得分条:每列下方会出现一个“得分条”,表示该位点保守性强弱。
2.多重比对结果的统计分析
在比对结果窗口点击Tools→AlignmentStatistics;
可查看以下关键指标:
序列相似性百分比;
每一列的保守性得分;
插入/缺口统计;
平均对齐得分;
对不同区域(如N端、C端或结构域区域)还可局部统计。
3.标注结构域或功能区域
选中某条蛋白序列后,点击右键选择“AnnotateRegion”;
可添加如“KinaseDomain”、“ATPBindingSite”等结构域标注;
这些标注可在多重比对中直接显示,方便对比功能保持性。
4.比对结果导出
DNAMAN支持将比对结果导出为多种格式:
导出为FASTA:对齐后的序列保存为标准FASTA格式,方便其他软件继续分析;
导出为PDF或图片:在“File→ExportAlignmentView”中选择“Image”或“PDF”,用于论文或演示;
导出为Clustal格式:便于与MEGA、BioEdit、JalView等软件兼容使用;
导出为CSV:统计结果可另存为表格格式,用于报告编写。
5.构建系统发育树(进阶)
基于比对结果,点击Phylogeny→GenerateTree;
可生成NJ树(Neighbor-JoiningTree);
支持自定义树状图颜色、分支长度等;
树图可保存为图像,用于发表或讲座演示。
三、延伸技巧:如何优化多重蛋白比对质量
进行蛋白多重比对不仅仅是“点几下”这么简单,若想得到高质量的结果,更需掌握以下技巧:
1.精选序列输入源
保证输入序列长度相近,避免过短或冗长的序列干扰;
使用NCBI或UniProt中经过人工注释的高质量序列。
2.合理选择打分矩阵
BLOSUM62适用于一般同源分析;
PAM250适合分析进化距离较远的序列;
选择错误的矩阵可能导致错配位点增多。
3.控制Gap惩罚参数
对于高度保守序列,适当提高Gap开罚分;
若希望捕捉结构重排或插入片段,可降低Gap惩罚以增加敏感度。
4.局部比对与全长比对结合
有时全长蛋白比对会因两端非保守区拉低得分;
可只比对结构域区域或功能片段,以提高实际生物学意义。
5.使用一致性掩码
DNAMAN支持设置“ConsensusMask”,即隐藏低一致性区域;
适用于构建高质量保守性Logo图或功能位点可视化。
总结
本文围绕“DNAMAN如何比对蛋白DNAMAN怎么多重比对结果”展开了详尽的操作指导和分析技巧。从序列导入、参数配置,到比对结果可视化、导出与统计分析,DNAMAN凭借其清晰直观的操作界面和强大的数据处理能力,依然是当前中小型实验室进行蛋白比对的有力工具。掌握这些实用操作,不仅能提高你的数据处理效率,也为后续的功能注释、突变分析或系统发育研究打下坚实的基础。

