在分子生物学研究中,DNA或蛋白质序列比对是非常常见的工作内容,而一款操作直观、功能实用的工具能够大大提高实验效率。DNAMAN是一款经典的序列分析软件,广泛用于核酸与蛋白序列的编辑、比对、翻译、注释等工作。无论是对单个基因片段的核酸序列对齐,还是多个蛋白质序列的系统发育分析,DNAMAN都能提供简洁清晰的可视化结果。本文将围绕两个方面详细介绍:DNAMAN序列比对步骤以及DNAMAN序列比对结果怎么看,帮助新手快速掌握该工具的基本用法。
一、DNAMAN序列比对步骤
DNAMAN支持包括ClustalW、Pairwise等多种比对方式。进行序列比对前,用户需要先准备好待比对的FASTA格式序列或通过软件界面直接粘贴输入。以下为常规使用流程:
1.导入序列
打开DNAMAN后,可通过以下两种方式导入序列:
方法一:点击菜单栏的“File”→“Open”,选择已保存的.seq或.fasta文件。
方法二:新建一个文件,将多条序列复制粘贴至软件界面中,再依次保存为不同的序列条目。
确认每条序列格式正确后,保存成项目文件,以便统一管理。
2.进行多序列比对
比对过程如下:
依次选中需要比对的两个或多个序列(按住Ctrl键可多选)。
点击菜单栏“Align”→选择“MultipleSequenceAlignment”。
在弹出的参数设置中选择比对方式(如ClustalW、Globalalignment)、GapPenalty(空位惩罚)、延伸惩罚等。
确认后点击“OK”开始运行比对。
完成比对后,会弹出一个新的窗口,展示所有序列的对齐结果。
3.可视化设置与保存
你可以根据需要设置比对颜色风格,如:
显示保守位点(用“*”表示)
高亮相同碱基或氨基酸
使用Shading显示差异等级
最后点击“File”→“Saveas”保存比对结果为.msf或.rtf格式,便于后续汇报、投稿或图表处理。
二、DNAMAN序列比对结果怎么看
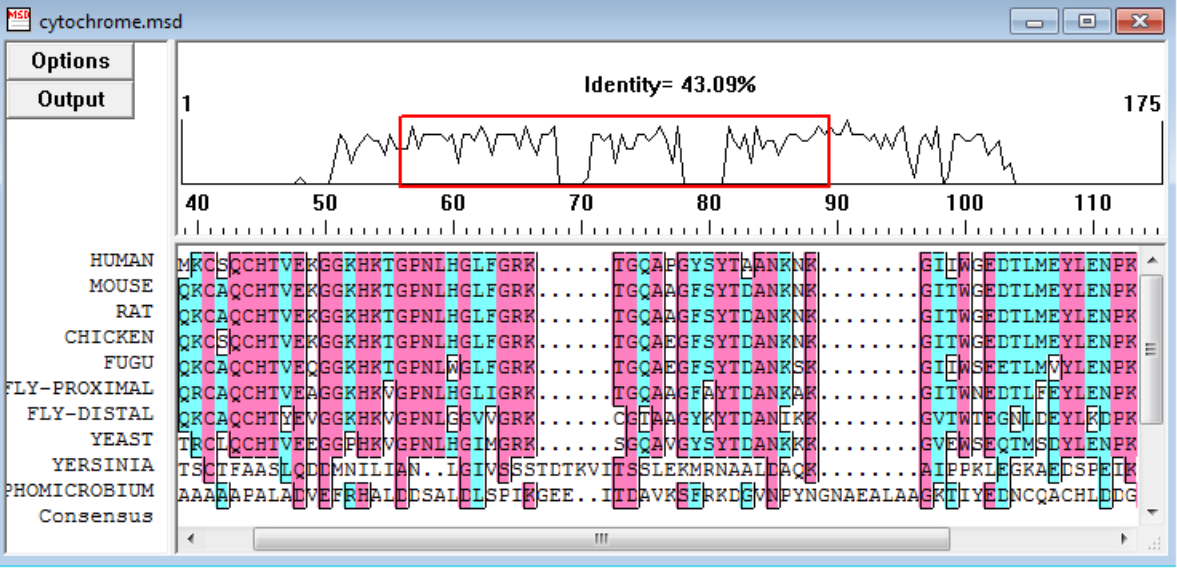
完成比对后,我们要学会如何解读比对图,以便判断序列之间的相似性、变异位点和进化关系。DNAMAN的比对结果窗口通常包括以下几个核心部分:
1.比对视图结构
序列列表:左侧为每条参与比对的序列名称。
主比对区域:每列代表同一位置上各序列的字符(碱基或氨基酸)。
保守性标注:
*表示该位置所有序列完全一致(高度保守)
:表示该位置为相似氨基酸(中度保守)
.表示弱相似区域
空格表示不保守或有缺失
通过这些符号,我们可以一目了然地看出序列的相似度。
2.颜色编码与阴影辅助理解
默认情况下,DNAMAN会用不同颜色标出相同或相似的位点,如A/T/G/C或不同类型的氨基酸分类。如果你开启了“ConservationShading”,高保守性区域会被染成深色,便于重点分析。
你还可以通过设置“ColorbyIdentity”、“ColorbyGroup”来更直观地区分亲缘关系。
3.常见结果应用场景
构建系统发育树:从比对结果中输出.phy或.nexus文件供MEGA或PhyML使用。
突变识别:对比样本序列与参考序列,寻找SNP或缺失位点。
功能位点比较:找出功能保守的关键氨基酸位点是否发生突变。
比对结果不仅用于比对本身,更是后续功能注释、进化分析和分子标记开发的重要基础。
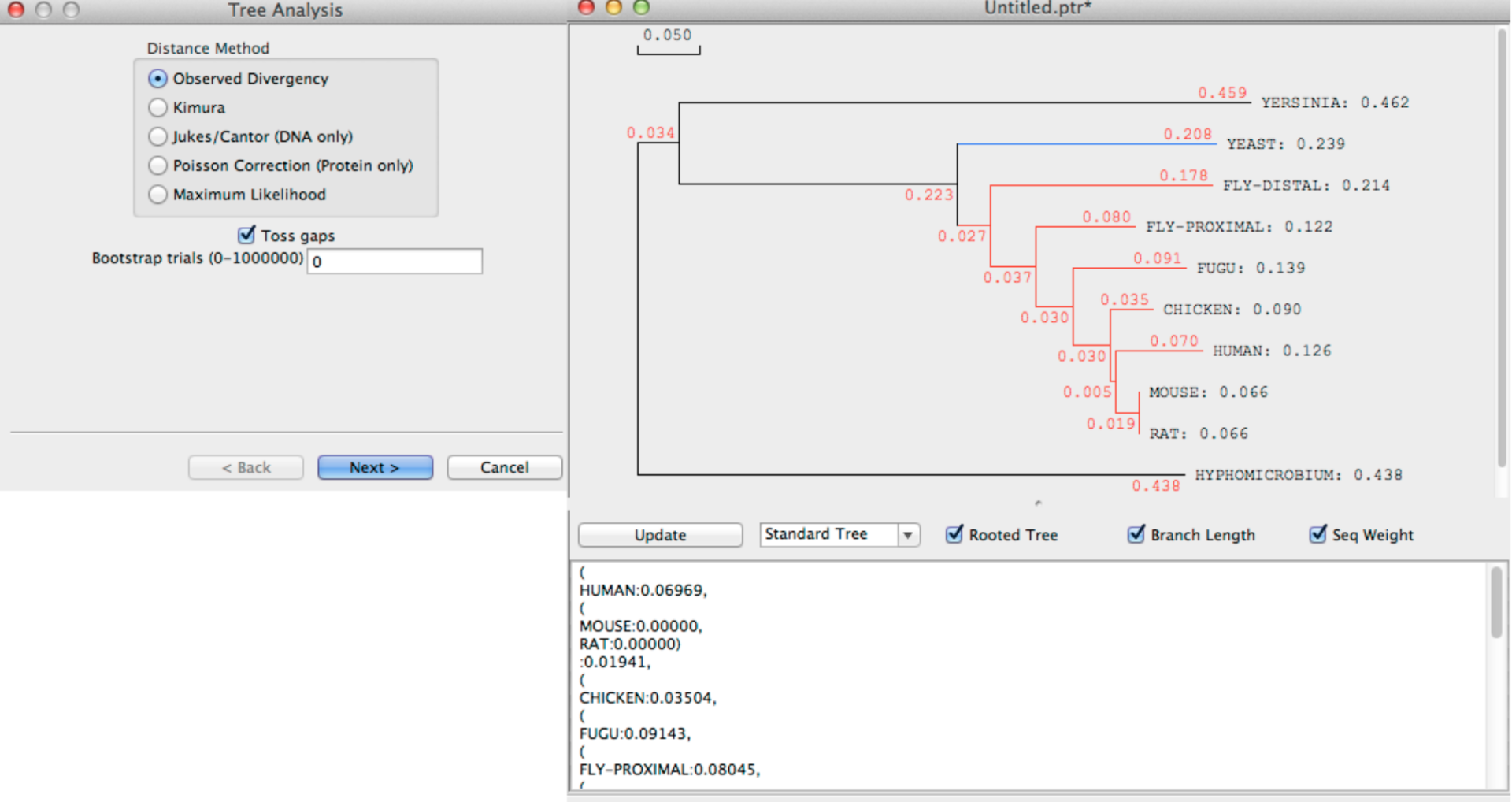
三、DNAMAN比对结果如何用于进化分析和科研发表
在科研工作中,比对并不仅仅是看“有没有差异”,更重要的是将这些差异具象化、解释化、数据化,以用于后续分析和成果输出。
1.构建系统发育树
DNAMAN本身支持初步的树形图显示,但通常建议将比对结果导出为.aln或.phy格式,导入MEGA、RAxML或PhyML等工具进行更精细的系统发育树构建。
2.数据整理用于图表输出
DNAMAN的比对图可以直接导出为RTF格式,在Word或PPT中清晰展示序列结构对比,是论文投稿中常见的图表素材之一。也可以截图高保守区域作为图注素材。
3.配合突变注释数据库进行功能关联分析
当你识别出关键差异位点后,可结合UniProt、NCBI、Pfam等数据库检索该位点的生物学功能,进一步探究其对蛋白功能或表型的影响,从而挖掘研究亮点。
总结
综上所述,DNAMAN序列比对步骤DNAMAN序列比对结果怎么看涵盖了从序列导入、比对设定、结果读取到可视化输出的完整操作流程。作为一款经典的生物信息学分析工具,DNAMAN上手简单,界面直观,特别适合初学者和日常科研人员快速进行核酸或蛋白序列比对分析。掌握其比对逻辑和结果解读方法,不仅能提升分析效率,也为后续论文撰写和项目设计打下坚实基础。

